1 Beh.faktor - Split-Plot Design
Datensatz
library(data.table) # bessere Datenmanipulation
library(ggplot2) # bessere Plots
library(emmeans) # adjustierte Mittelwerte
library(lme4); library(lmerTest) # gemischtes Modell
library(RColorBrewer) # bessere FarbenIn diesem Experiment wurden die Auswirkungen von 6 Stickstoffdüngern (N) auf den Ertrag von 4 Reissorten (G) untersucht. Demnach gab es zwei Behandlungsfaktoren mit insgesamt 6x4=24 Behandlungsfaktorstufenkombinationen. Diese 24 Kombinationen wurden 3 mal in vollständigen Blöcken wiederholt. Innerhalb der vollständigen Blöcke wurden die Kombinationen allerdings nicht wie bei einem RCBD vollständig randomisiert. Grund hierfür war, dass es beim Anlegen des Feldversuchs und speziell beim Ausbringen der Dünger praktischer ist, wenn die Parzellen mit demselben Dünger in einer Fahrtrichtung liegen. Deshalb wurden die vollständigen Blöcke erst in 6 Spalten (=main plots) unterteilt, auf welche die 6 N-Stufen randomisert wurden. Erst dann, wurden die 4 Sorten den 4 Zeilen innerhalb jeder Spalte (=sub plots) zufällig zugeordnet. Solch eine Versuchsanlage nennt man split-plot design. Hier mehr Infos zu Versuchsdesigns
## Warning in if (class(data) == "formula") {: the condition has length > 1 and only
## the first element will be used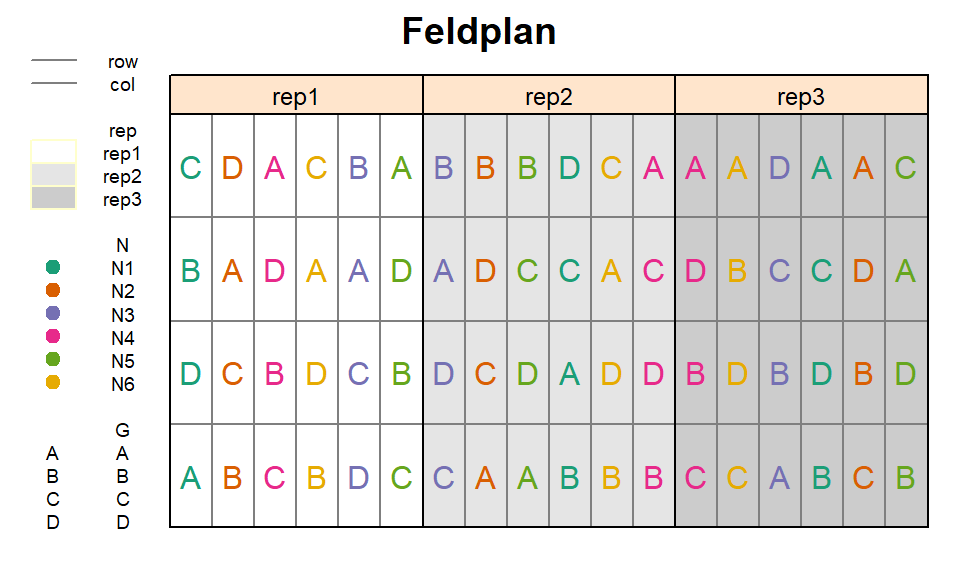
print(splitplot, nrows=10)## N G rep mainplot yield
## 1: N1 A rep1 mp1 4520
## 2: N2 A rep1 mp2 5598
## 3: N4 A rep1 mp3 6192
## 4: N6 A rep1 mp4 8542
## 5: N3 A rep1 mp5 5806
## ---
## 68: N6 D rep3 mp14 1744
## 69: N3 D rep3 mp15 4236
## 70: N1 D rep3 mp16 5016
## 71: N2 D rep3 mp17 4382
## 72: N5 D rep3 mp18 2856str(splitplot)## Classes 'data.table' and 'data.frame': 72 obs. of 5 variables:
## $ N : Factor w/ 6 levels "N1","..
## $ G : Factor w/ 4 levels "A",""..
## $ rep : Factor w/ 3 levels "rep1"..
## $ mainplot: Factor w/ 18 levels "mp1"..
## $ yield : int 4520 5598 6192 8542 ..
## - attr(*, ".internal.selfref")=<exter..Deskriptive Statistik
Erst wollen wir ein Gefühl für den Datensatz bekommen und betrachten einen Boxplot für die Stufenkombinationen der zwei Behandlungsfaktoren. Mittels des col= statements der boxplot() Funktion können wir angeben welche Füllfarben die Boxen haben sollen. Wir wählen dieselben Farben wie im Feldplan - mehr Infos dazu hier.
boxplot(yield ~ N*G, col=brewer.pal(6, "Dark2"), data=splitplot, las=2) # las=2 dreht Achsenbeschriftung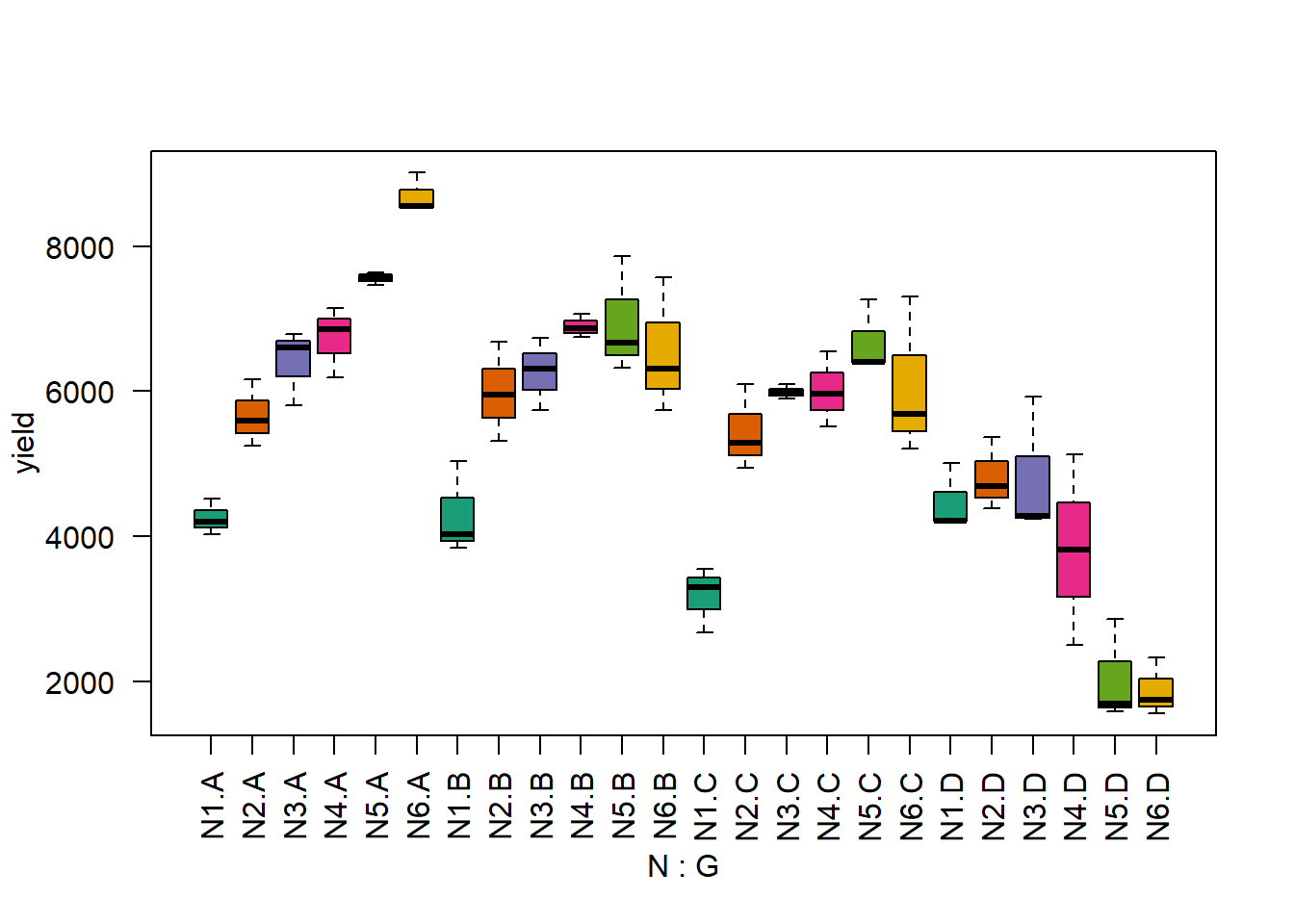
Es fällt auf, dass sich das Muster der Werte der verschiedenen N-Stufen für Sorte D deutlich von denen der anderen 3 Sorten unterscheiden. Dies deutet auf Wechselwirkungen zwischen den beiden Behandlungsfaktoren hin.
Schließende Statistik
Gemischtes Modell
Wir können uns nun entschließen die Daten mittels eines Modells zu analysieren. Der Ertrag ist unsere metrische Zielvariable. ‘G’ und ‘N’ sind beides qualitative Behandlungsfaktoren. Um eine möglich Wechselwirkung der beiden zu modellieren, ergibt sich der treament part des Modell als G + N + G*N. Für die vollständigen Blöcke haben wir den festen Effekt ‘rep’ im Modell. Desweiteren müssen wir zufällige Effekte für die main plots ins Modell aufnehmen, da diese als zusätzliche Randomisationseinheit fungierten. Die andere, kleinste Randomisationseinheit des sub plots (=Parzelle) wird durch den üblichen Fehlerterm abgebildet. Der design part des Modells ist also rep + (1|rep:mainplot). Somit haben wir gleichzeitig feste und zufällige Effekte im Modell und demnach ein gemischtes Modell, welches wir wieder mit lmer() aus dem lme4 package modellieren.
mod <- lmer(yield ~ G + N + G:N + rep + (1|rep:mainplot), data=splitplot)Zunächst sollten nun die Residuenplots evaluiert werden - beispielsweise mit plot(mod) und qqnorm(resid(mod)); qqline(resid(mod)). Erst danach ist eine Varianzanalyse zulässig.
Die Spalte ‘mainplot’ ist in diesem Beispiel übrigens prinzipiell überflüssig. Das liegt daran, dass man die 3x6 main plots anstatt mit ‘rep:mainplot’ auch mit ‘rep:N’ hätte modellieren können, da ein main plot immer einer N-Stufe entspricht (siehe Feldplan oben). Um es aber leichter verständlich zu machen, nutzen wir hier eine extra ‘mainplot’ Spalte. Siehe außerdem die Anmerkung aus dem vorigen Beispiel.
Varianzanalyse
library(car)
Anova(mod, test.statistic="F", type="III")## Analysis of Deviance Table (Type III Wald F tests with Kenward-Roger df)
##
## Response: yield
## F Df Df.res Pr(>F)
## (Intercept) 115.2209 1 37.499 5.430e-13 ***
## G 3.0166 3 36.000 0.0424 *
## N 17.5989 5 41.916 2.293e-09 ***
## rep 0.9774 2 10.000 0.4095
## G:N 13.2360 15 36.000 2.078e-10 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Da dies Ergebnisse einer mehrfaktorielle Varianzanalyse sind, betrachten zuallererst den F-Test für die Wechselwirkung (mehr Infos hier). Der signifikante p-Wert von G:N deutet darauf hin, dass die beiden Behandlungsfaktoren nicht rein additiv wirken, sondern wechselwirken. Dies bestätigt die Vermutung aus dem Boxplot der Rohdaten oben. Das bedeutet außerdem, dass wir die adjustierten Mittelwerte der Wechselwirkungseffekte berechnen wollen und nicht die der beiden Haupteffekte.
Multipler Mittelwertvergleich
Mit emmeans() können wir auch adjustierte Mittelwerte für Wechselwirkungseffekte berechnen. Das geht entweder mit pairwise ~ N : G oder pairwise ~ N | G. Im beiden Fällen werden wie erwartet Mittelwerte für alle Kombinationen errechnet. Der Unterschied zwischen den beiden Befehlen besteht darin welche Differenzen zwischen den Mittelwerten berechnet werden. Im ersten Fall (N:G) werden alle Mittelwerte miteinander verglichen. Im zweiten Fall (N|G), werden nur N-Stufen innerhalb einer Sorte miteinander verglichen. Natürlich können ‘N’ und ‘G’ hier auch ausgetauscht werden. Es kommt auf die Versuchsfrage an, welche Mittelwertvergleiche von Interesse sind. Wir wollen uns hier auf die Vergleiche der N-Mittelwerte getrennt pro Sorte fokussieren.
means <- emmeans(mod, pairwise ~ N | G, adjust="tukey") # adjust="none" für t-test
means <- CLD(means$emmeans, details=TRUE, Letters=letters)## Warning: 'CLD' will be deprecated. Its use is discouraged.
## See '?cld.emmGrid' for an explanation. Use 'pwpp' or 'multcomp::cld' instead.means$emmeans # N-Mittelwerte pro Sorte## G = A:
## N emmean SE df lower.CL upper.CL .group
## N1 4252.667 365.5222 41.92 3243.437 5261.896 a
## N2 5672.000 365.5222 41.92 4662.771 6681.229 ab
## N3 6400.000 365.5222 41.92 5390.771 7409.229 bc
## N4 6732.667 365.5222 41.92 5723.437 7741.896 bc
## N5 7563.333 365.5222 41.92 6554.104 8572.563 cd
## N6 8700.667 365.5222 41.92 7691.437 9709.896 d
##
## G = B:
## N emmean SE df lower.CL upper.CL .group
## N1 4306.000 365.5222 41.92 3296.771 5315.229 a
## N2 5982.000 365.5222 41.92 4972.771 6991.229 b
## N3 6259.000 365.5222 41.92 5249.771 7268.229 b
## N6 6540.333 365.5222 41.92 5531.104 7549.563 b
## N4 6895.000 365.5222 41.92 5885.771 7904.229 b
## N5 6950.667 365.5222 41.92 5941.437 7959.896 b
##
## G = C:
## N emmean SE df lower.CL upper.CL .group
## N1 3177.333 365.5222 41.92 2168.104 4186.563 a
## N2 5442.667 365.5222 41.92 4433.437 6451.896 b
## N3 5994.000 365.5222 41.92 4984.771 7003.229 b
## N4 6014.000 365.5222 41.92 5004.771 7023.229 b
## N6 6065.333 365.5222 41.92 5056.104 7074.563 b
## N5 6687.333 365.5222 41.92 5678.104 7696.563 b
##
## G = D:
## N emmean SE df lower.CL upper.CL .group
## N6 1880.667 365.5222 41.92 871.437 2889.896 a
## N5 2046.667 365.5222 41.92 1037.437 3055.896 a
## N4 3816.000 365.5222 41.92 2806.771 4825.229 b
## N1 4481.333 365.5222 41.92 3472.104 5490.563 b
## N3 4812.000 365.5222 41.92 3802.771 5821.229 b
## N2 4816.000 365.5222 41.92 3806.771 5825.229 b
##
## Results are averaged over the levels of: rep
## Degrees-of-freedom method: kenward-roger
## Confidence level used: 0.95
## Conf-level adjustment: sidak method for 6 estimates
## P value adjustment: tukey method for comparing a family of 6 estimates
## significance level used: alpha = 0.05means$comparisons # N-Mittelwertdifferenzen pro Sorte## G = A:
## contrast estimate SE df t.ratio p.value
## N2 - N1 1419.333 516.9265 41.92 2.746 0.0873
## N3 - N1 2147.333 516.9265 41.92 4.154 0.0020
## N3 - N2 728.000 516.9265 41.92 1.408 0.7218
## N4 - N1 2480.000 516.9265 41.92 4.798 0.0003
## N4 - N2 1060.667 516.9265 41.92 2.052 0.3317
## N4 - N3 332.667 516.9265 41.92 0.644 0.9869
## N5 - N1 3310.667 516.9265 41.92 6.405 <.0001
## N5 - N2 1891.333 516.9265 41.92 3.659 0.0086
## N5 - N3 1163.333 516.9265 41.92 2.250 0.2373
## N5 - N4 830.667 516.9265 41.92 1.607 0.5987
## N6 - N1 4448.000 516.9265 41.92 8.605 <.0001
## N6 - N2 3028.667 516.9265 41.92 5.859 <.0001
## N6 - N3 2300.667 516.9265 41.92 4.451 0.0008
## N6 - N4 1968.000 516.9265 41.92 3.807 0.0057
## N6 - N5 1137.333 516.9265 41.92 2.200 0.2593
##
## G = B:
## contrast estimate SE df t.ratio p.value
## N2 - N1 1676.000 516.9265 41.92 3.242 0.0264
## N3 - N1 1953.000 516.9265 41.92 3.778 0.0061
## N3 - N2 277.000 516.9265 41.92 0.536 0.9943
## N6 - N1 2234.333 516.9265 41.92 4.322 0.0012
## N6 - N2 558.333 516.9265 41.92 1.080 0.8865
## N6 - N3 281.333 516.9265 41.92 0.544 0.9939
## N4 - N1 2589.000 516.9265 41.92 5.008 0.0001
## N4 - N2 913.000 516.9265 41.92 1.766 0.4977
## N4 - N3 636.000 516.9265 41.92 1.230 0.8195
## N4 - N6 354.667 516.9265 41.92 0.686 0.9825
## N5 - N1 2644.667 516.9265 41.92 5.116 0.0001
## N5 - N2 968.667 516.9265 41.92 1.874 0.4319
## N5 - N3 691.667 516.9265 41.92 1.338 0.7624
## N5 - N6 410.333 516.9265 41.92 0.794 0.9671
## N5 - N4 55.667 516.9265 41.92 0.108 1.0000
##
## G = C:
## contrast estimate SE df t.ratio p.value
## N2 - N1 2265.333 516.9265 41.92 4.382 0.0010
## N3 - N1 2816.667 516.9265 41.92 5.449 <.0001
## N3 - N2 551.333 516.9265 41.92 1.067 0.8917
## N4 - N1 2836.667 516.9265 41.92 5.488 <.0001
## N4 - N2 571.333 516.9265 41.92 1.105 0.8765
## N4 - N3 20.000 516.9265 41.92 0.039 1.0000
## N6 - N1 2888.000 516.9265 41.92 5.587 <.0001
## N6 - N2 622.667 516.9265 41.92 1.205 0.8322
## N6 - N3 71.333 516.9265 41.92 0.138 1.0000
## N6 - N4 51.333 516.9265 41.92 0.099 1.0000
## N5 - N1 3510.000 516.9265 41.92 6.790 <.0001
## N5 - N2 1244.667 516.9265 41.92 2.408 0.1769
## N5 - N3 693.333 516.9265 41.92 1.341 0.7606
## N5 - N4 673.333 516.9265 41.92 1.303 0.7819
## N5 - N6 622.000 516.9265 41.92 1.203 0.8328
##
## G = D:
## contrast estimate SE df t.ratio p.value
## N5 - N6 166.000 516.9265 41.92 0.321 0.9995
## N4 - N6 1935.333 516.9265 41.92 3.744 0.0068
## N4 - N5 1769.333 516.9265 41.92 3.423 0.0164
## N1 - N6 2600.667 516.9265 41.92 5.031 0.0001
## N1 - N5 2434.667 516.9265 41.92 4.710 0.0004
## N1 - N4 665.333 516.9265 41.92 1.287 0.7903
## N3 - N6 2931.333 516.9265 41.92 5.671 <.0001
## N3 - N5 2765.333 516.9265 41.92 5.350 <.0001
## N3 - N4 996.000 516.9265 41.92 1.927 0.4008
## N3 - N1 330.667 516.9265 41.92 0.640 0.9872
## N2 - N6 2935.333 516.9265 41.92 5.678 <.0001
## N2 - N5 2769.333 516.9265 41.92 5.357 <.0001
## N2 - N4 1000.000 516.9265 41.92 1.935 0.3963
## N2 - N1 334.667 516.9265 41.92 0.647 0.9865
## N2 - N3 4.000 516.9265 41.92 0.008 1.0000
##
## Results are averaged over the levels of: rep
## Degrees-of-freedom method: kenward-roger
## P value adjustment: tukey method for comparing a family of 6 estimatesErgebnisaufbereitung
Wir wollen die Ergebnisse abschließend in einem Balkendiagramm darstellen. Weil wir aber die N-Vergleiche isoliert pro Sorte konzentrieren wollen, können wir uns entscheiden ein Balkendiagramm pro Sorte zu erstellen. In solch einer Situation kann in der ggplot() Funktion das facet_wrap() statement genutzt werden.
Außerdem wollen wir die Balken wieder mit denselben Farben versehen, die die N-Stufen bereits im Feldplan und dem Boxplot hatten. Dazu fügen wir zuerst dem geom_bar() statement fill=N hinzu. Nun bekommt jede N-Stufe eine andere Farbe. Um nicht die default Farben zu nehmen, sondern selbst zu bestimmen welche Farben es sein sollen, schreiben wir zusätzlich noch scale_fill_manual(values=...) in die Funktion. Um die dann automatisch generierte Legende zu unterdrücken (weil sie in diesem Fall keine zusätzlichen Informationen bringt), fügen wir außerdem guides(fill=FALSE) hinzu.
means.plot <- as.data.table(means$emmeans) # korrektes Format für ggplot
print(means.plot, nrows=2)## N G emmean SE df lower.CL upper.CL .group
## 1: N1 A 4252.667 365.5222 41.91628 3243.437 5261.896 a
## 2: N2 A 5672.000 365.5222 41.91628 4662.771 6681.229 ab
## 3: N3 A 6400.000 365.5222 41.91628 5390.771 7409.229 bc
## 4: N4 A 6732.667 365.5222 41.91628 5723.437 7741.896 bc
## 5: N5 A 7563.333 365.5222 41.91628 6554.104 8572.563 cd
## ---
## 20: N5 D 2046.667 365.5222 41.91628 1037.437 3055.896 a
## 21: N4 D 3816.000 365.5222 41.91628 2806.771 4825.229 b
## 22: N1 D 4481.333 365.5222 41.91628 3472.104 5490.563 b
## 23: N3 D 4812.000 365.5222 41.91628 3802.771 5821.229 b
## 24: N2 D 4816.000 365.5222 41.91628 3806.771 5825.229 bggplot(data=means.plot, aes(x=N)) +
geom_bar(aes(y=emmean, fill=N), stat="identity", width=0.8) +
scale_fill_manual(values=brewer.pal(6, "Dark2")) + guides(fill=FALSE) +
geom_errorbar(aes(ymin=lower.CL, ymax=upper.CL), width=0.4) +
geom_text(aes(y=emmean+1500, label=.group)) +
facet_wrap(~G) + # ein plot pro Sorte
theme_bw() +
labs(main="Adj. N-Mittelwerte pro Sorte",
y="Adjustierter Ertragsmittelwert ± 95% Konfidenzintervall", x="N-Dünger",
caption="Getrennt für jede Sorte gilt: Mittelwerte, die mit einem gemeinsamen Buchstaben versehen sind,\n sind laut Tukey-test nicht signifikant voneinander verschieden.")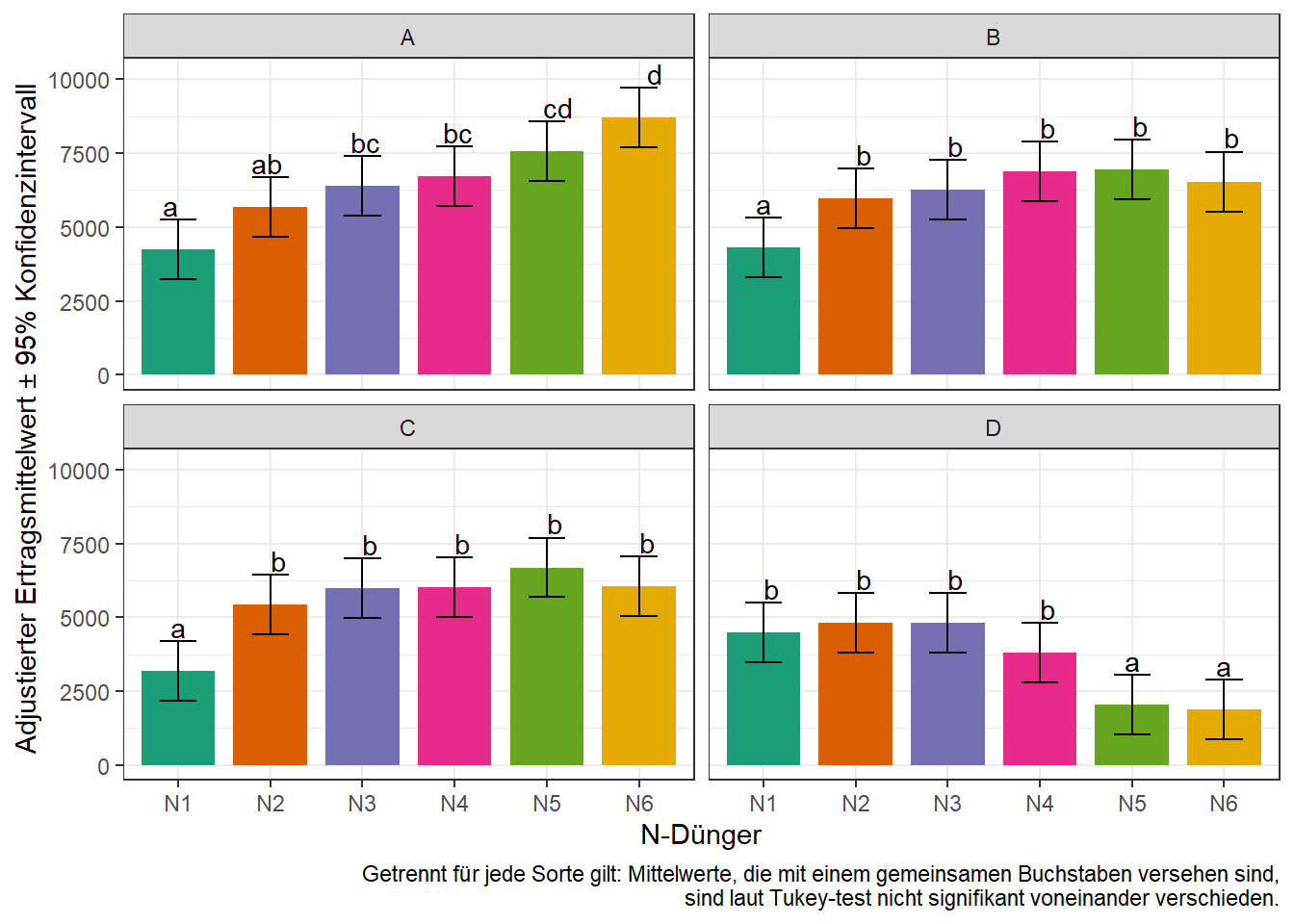
Bei Fragen kannst du mir gerne schreiben!
schmidtpaul@hotmail.de